See the protocol here: https://www.protocols.io/view/growing-bacteria-in-superbroth-thick-food-3qmgmu6
Dans cette catégorie, vous trouverez des protocoles de laboratoire, accompagnés le plus souvent d’images et de vidéos.
See the protocol here: https://www.protocols.io/view/growing-bacteria-in-superbroth-thick-food-3qmgmu6
<!doctype html>
DNA Purification
Defrost the stock into an agar plate.
The strains I am working with should be isogenic but to increase the probability of working with an isogenic strain, I created
a new strain originating from a single worm. Pick a single worm into a new fresh agar NGM plate. Let it move away from the point
of deposition. Pick it again into a new fresh plate. This procedure of first picking into a plate and then into another makes sure
that no other worms or eggs are co-picked with the single worm of interest. Let the single worm populate the whole plate and
chunk the plate into 2 or more plates. This strain is now called differently, starting with SX, the code given to the Miska lab.
Freeze of of the plates.
Always freeze 5 vials. White plug in one of the 5 red caps. New strain from single worm is called
SX…. . SX is the Miska lab designation in the world. One of the tubes will be thawed tomorrow to check
that the freezing worked. One tube will be put in liquid nitrogen for long term storage. Three tubes will be
put in the -80 C freezer for mid term storage. Take a 15 ml Falcon tube, M9 medium, freezing buffer 2X.
Final volume of vial: 1 ml, so need 2.5 ml of freezing buffer. Pour 3.2 ml M9 on plate. Take 2.5 ml back with
pipetboy and 5 ml serological pipette. Put in Falcon. Add 2.5 ml freezing buffer to falcon. Pipet 1 ml of
solution in each vial. Transfer the vials into a polystyrene box. Tape the polystyrene box shut. Put the polystyrene box
in the -80 C freezer. The box allows the temperature decrease to be slower. Tomorrow, put test tube in tap water for 5 min.
Pour on plate and check that worms are alive. Put other tubes in inbox (4 tubes) and email lab manager. New strain SX…. is ready.
Pick a few worms into a fresh agar plate.
Once many gravid adults are present on the plate, bleach them and grow the eggs overnight at -20 C on a rotating wheel.
Prepare 50 ml of bleach solution in a 50 ml Falcon tube: 4mL 10M NaOH, 3mL Sodium hypochlorite (stored in the cold room), 43mL H2O. The bleach solution can be stored at room temperature for one month.
Tip: Pipette gently to avoid collecting bits of agar together with the worms.
Measure the nucleic acid concentration using Nanodrop.
Lift pedestal.
Clean sample area.
Drop pedestal.
Touch screen.
-> Nucleic Acids -> dsDNA
Instrument self-test…
Lift pedestal.
Clean pedestal.
Load blank.
Lower arm.
Export Nanodrop results onto USB stick.
Measure the dsDNA concentration using Qubit.
Use low sensitivity dye (BR: broad range) dilute 200x reagent in buffer to give working solution prepare two
standards tubes in Qubit tubes
Agarose gel: 250 ml flask. Add 0.45 g agarose in 60 ml TAE in measuring cylinder.
Tape container. Put comb in container. Microwave 1 min. Add 0.6 ul ETBR to solution.
Stir. Pour in 4 C room. Add water in flask. Stir. Pour through white material for ETBR. Wash flask with water.
Remove tape. Put gel in container. Add 5 ul ladder. Add 2 ul loading buffer to 5 ul DNA sample and load into gel.
Turn on machine on right. 150 mV, 400 mA, 30 min. Check run every 10 minutes.
Put gel in Imager. Open Image Lab. New Protocol.
Position gel, zoom in/out. Start Protocol. Image is ready on computer.
Invert contrast, change contrast, print image. Don’t touch the computer without gloves. It is contaminated.
Thanks to Jérémie Le Pen and others from the Miska lab for the invaluable advice.
DO NOT ADD THE KCl TO THE AGAROSE SOLUTION BEFORE MICROWAVING THE AGAROSE!!! (COULD CAUSE SPARKS IF KCl IS MICROWAVED)
Capillaries should not contain any bubbles.
Store the capillaries, i.e. agar bridges, at room temperature in a 3 M KCl solution.
Matrigel 6 cm diameter Petri dishes:
During the incubation, trypsinise a T75 flask of GLUTag cells and resuspend them in 10 ml medium (50/50 fresh/conditioned media).
Remove the matrigel from the dish and add 4 ml full DMEM medium.
Add 2.5 ml of the resuspension in the dish and incubate overnight at 37°C/5% CO2.
This step is necessary in order to go from a confluent layer of cells to single cells scattered on a dish that are amenable to being patch-clamped.
Each dish contains 0.5 x 2/50 = 0.5 x 1/25 = 0.5 x 0.04 = 0.02 = 2 % of the initial cells. It is advisable to make another two-fold dilution to get 1% of the initial cells on a few dishes in case the former dilution is not strong enough.
Starting point: confluent T75 flask containing stuck GLUTag cells and 20 ml DMEM medium
All steps are undertaken in a hood!
Exchange 10 ml supernatant for 10 ml fresh DMEM medium
Refer to Day 1.
Cristian Riccio, Institute of Metabolic Science, University of Cambridge
Thanks to Dr. Edward Emery and Dr. Frank Reimann from the Institute of Metabolic Science for their contribution to the establishment of this protocol.
Autoclave 2 x 200 ml LB medium.
In the evening, inoculate the medium (containing the appropriate antibiotic) with either a bacterial colony or a chunk of glycerol stock. Grow overnight at 37°C with shaking at 220 rpm.
Refer to protocol for purification of native protein.
Starting point: T75 flask with GLUTag cells at a confluence > 80%
End point: 10 cm diameter Petri dish with GLUTag cells
Starting point:
End point:
pDNA stock concentration: 2 ug/ul
Starting point: Petri dish with transfected cells
End point: 24-wells plate with transfected cells
Starting point: 24-well plates containing attached transfected and untransfected GLUTag cells
End point: Frozen supernatants from stimulated cells
Cristian Riccio, Laboratory of Prof. Gribble and Dr. Reimann, Institute of Metabolic Science
Lausanne, le 28 février 2014
Souvent, lorsqu’on travaille avec une nouvelle construction plasmidique (construct), nous voulons vérifier que les bactéries qui ont incorporé le plasmide produisent bel et bien la protéine dont le gène se trouve sur le plasmide. Pour cela, il est conseillé d’effectuer un test d’expression de protéine à petite échelle avant de passer à une échelle plus grande, nécessaire à la production d’une quantité importante de protéine. Dans le cas où la protéine n’est pas exprimée, cela peut indiquer un problème avec le plasmide ou bien que la protéine est dégradée (en particulier dans le cas d’une expression dans des cellules eucaryotes, où le protéasome dégrade les protéines mal pliées) et il faut résoudre ce problème avant de passer à une expression à grande échelle.
Point de départ de l’expérience: colonies sur boîte d’agar.
Point d’arrivée: gels de polyacrylamide produits par SDS-PAGE et indiquant le degré d’expression de la protéine.
Marche à suivre
1. Choisir une ou plusieurs colonies en essayant d’éviter les colonies satellites (cf. lexique pour définition). Choisir plusieurs colonies augmente les chances d’obtenir au moins un résultat positif. Les étapes suivantes sont décrites pour une colonie.
2. Inoculer 4 tubes de culture de 12 ml contenant 2 ml de milieu de culture LB + antibiotique à partir d’une seule colonie. En outre, il est conseillé d’avoir un contrôle négatif et un contrôle positif pour l’expression. Pour le contrôle positif, utiliser un stock de glycérol dont on sait qu’il exprime une protéine (n’importe laquelle) et pour le contrôle négatif utiliser des bactéries non transformées. Enfin, utiliser un tube supplémentaire inoculé avec n’importe quelle colonie. Ce tube servira à mesur la densité optique (DO) à 600 nm.
3. Incuber avec rotation à 37°C pendant quelques heures jusqu’à ce que la densité optique à 600 nm du tube supplémentaire atteigne au moins 0.6. A ce moment-là, les 4 tubes inoculés avec la même colonie prennent des chemins différents:
a. Un tube, marqué iNI37, reste à 37°C sans aucun traitment. i va de 1 au nombre de colonies sélectionnées.
b. Un autre tube, marqué iI37, est induit à l’IPTG (1 mM de concentration finale) et reste à 37°C.
c. Un troisième tube, marqué iNI18, est déplacé à 18°C.
d. Le dernier tube, enfin, marqué iI18, est induit avec de l’IPTG et déplacé à 18°C. C’est ce dernier tube qui donne normalement l’expression maximale, en raison de l’induction et du déplacement à 18°C. A cette dernière température, les bactéries cessent de se multiplier et peuvent focaliser leur énergie sur la production de la protéine d’intérêt.
4. Laisser incuber au moins 3,5 heures aux températures respectives. Si c’est très tard et que vous voulez rentrer à la maison absolument, vous pouvez aussi laisser incuber du jour au lendemain.
5. Après l’incubation, transférer 1 ml de culture dans un tube eppendorf de 1,5 ml.
6. Centrifuger 10 min à 15’000 rpm.
7. Jeter le surnageant.
8. Resolubiliser le culot dans 200 µl de BugBuster. Cette solution va lyser les cellules.
9. Centrifuger 10 min à 15’000 rpm.
10. Transférer le surnageant (la partie soluble) dans un autre eppendorf de 1,5 ml.
11. Aliquoter 30 µl de partie soluble dans un autre tube eppendorf contenant 30 µl de solution Lämmli 2x (solution de chargement pour le SDS-PAGE).
12. Resuspendre le culot (la partie insoluble) dans 50 µl de solution Lämmli 2x.
13. Chauffer les tubes contenant le Lämmli à 95°C pendant 5 min et charger dans des gels de polyacrylamide.
Il est aussi possible d’utiliser des erlenmeyer au lieu des tubes de culture.
Lexique
Colonie satellite: colonie de bactéries non transformées qui réussit à pousser sur de l’agar contenant de l’antibiotique en raison de la dégradation de l’antibiotique par une colonie voisine qui exprime la résistance à l’antibiotique et dégrade l’antibiotique qui l’entoure.
Protocole (version courte en PDF)







Lausanne, le 27 février 2014
La transformation des bactéries avec un plasmide est utile si nous voulons exprimer un gène dans une souche de bactéries, par exemple un gène codant pour une protéine. Dans mon travail au Laboratoire de biologie structurale et biophysique à l’EPFL, je suis souvent amené à transformer des bactéries avec un plasmide. Aujourd’hui par exemple, j’aimerais transformer 4 plasmides contenant chacun le gène de la protéine X avec une mutation différente. J’aimerais transformer ces plasmides dans la souche E. coli B834(De3) parce que cette dernière se prête particulièrement bien à l’expression de protéines. J’utilise les bactéries en quelque sorte comme des petites usines pour produire ma protéine d’intérêt. D’autres souches de E. coli se prêtent mieux à d’autres fins, e.g. la souche DH5α se prête bien à la production de plasmide afin de l’extraire par préparation de plasmide (miniprep, midiprep ou maxiprep) pour augmenter notre stock de plasmide.
Aujourd’hui, je procèderai à une transformation chimique des bactéries. Il existe aussi la transformation électrique (par électroporation) des bactéries, discutée dans un autre post.
Marche à suivre
1. Sortir les bactéries compétentes B834(De3) du congélateur à – 80 °C et les faire dégeler sur de la glace. Faire de même avec les plasmides stockés à – 20 °C.
2. Pour chaque transformation, transférer 50 µl de bactéries compétentes dans un tube eppendorf de 1,5 ml.
3. Ajouter 2 µl de plasmide à une concentration de 15 ng/µl. La quantité finale de plasmide doit se situer entre 10 ng et 100 ng. Dans le cas présent, la mass finale de plasmide est de 30 ng.
4. Incuber le mélange de plasmide et de bactéries compétentes pendant 30 minutes sur la glace.
5. Pendant les 30 minutes d’incubation, nous allons préparer des boîtes d’agar contenant de l’antibiotique (ici de l’ampicilline). Si les boîtes sont déjà prêtes, les sortir du frigo et les mettre dans le thermostat à 37°C. Dans le cas contraire, nous allons préparer des boîtes d’agar fraîches. D’abord, faire fondre l’agar au micro-ondes. Perso, je fais fondre graduellement, i.e. pendant 2 minutes séparées d’un coup d’oeil pour monitorer l’avancement de la fusion et l’état du micro-ondes. Attention à que le bouchon ne soit pas fortement fermé (tight). Le bouchon doit pouvoir danser sur la bouteille, i.e. être lâche (loose), afin que l’air puisse sortir de la bouteille pendant l’augmentation de température (rappel de la loi des gaz parfaits: PV = nRT -> si la température augmente et que la bouteille est fermée (V constant), la pression dans la bouteille augmente et celle-ci risque d’exploser!). Après fusion de l’agar, en verser une quantité suffisante dans des tubes falcon de 50 ml (environ 20 ml par boîte de Pétri). Attendre que la température baisse suffisamment avant d’ajouter l’antibiotique. En effet, si l’antibiotique est ajouté trop tôt, la température élevée risque de le détruire. D’un autre côté, si nous attendons trop avant d’ajouter l’antibiotique, l’agar risque de se solidifier. Les étapes suivantes se passent près d’une flamme au bec Bunsen afin de ne pas contaminer l’agar avec des bactéries de l’air. Aujourd’hui, je prépare 5 boîtes d’agar avec antibiotique et je verse donc 100 ml d’agar dans 2 tubes falcons de 50 ml. Après que la température soit descendue suffisamment (un bon indice est qu’en touchant le tube avec l’avant bras, la sensation ne doit pas être douloureuse), j’ajoute 50 µl d’antibiotique 1000 x dans chaque tube. Je verse ensuite l’agar liquide dans les boîtes autour de la flamme et je les laisse ouvertes afin qu’elles sèchent.
6. Après les 30 minutes d’incubation sur glace, nous passons au choc thermique: chauffer les bactéries pendant 5 minutes à 42 °C sur un bloc chauffant.
7. Remettre les tubes sur glace pendant 2 minutes.
8. Ajouter 1 ml de milieu LB dans le tube eppendorf de 1,5 ml et transférer le tout (1,057 ml) dans des tubes de culture de 12 ml à incuber pendant 30-60 min à 37 °C à 220 rpm. Cette période de temps permet aux bactéries d’exprimer la résistance à l’antibiotique codée sur le plasmide.
9. Etaler 50 µl sur les boîtes d’agar contenant l’antibiotique. Seules les bactéries ayant incorporé l’antibiotique formeront des colonies pendant la nuit.
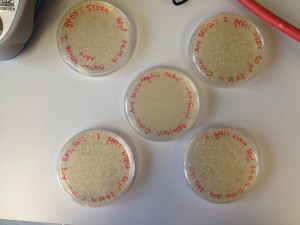
10. Incuber du jour au lendemain à 37°C dans le thermostat.
Lausanne, le 28 février 2014
9. Vérifier la présence de colonies dans les boîtes sur lesquelles des bactéries transformées ont été étalées. Vérifier que le contrôle négatif, la boîte sur laquelle des bactéries non transformées ont été étalées, ne contienne pas de colonies.
ingrédients
– des bactéries “compétentes”, i.e. facilement transformables par de l’ADN exogène. Les bactéries doivent soit être sensibles à un antibiotique, soit être auxotrophes.
– de l’agar en poudre
– un plasmide portant un gène de sélection. e.g. un gène produisant de la beta-lactamase donnant la résistance à l’antibiotique ampicilline ou un gène dont le produit permet la synthèse d’un nutriment rendant les bactéries qui l’expriment prototrophes.
– en fonction du gène de sélection: antibiotique ou milieu sélectif pour bactéries prototrophes.